1. Что значит код заданного фермента
Белок MLTA_ECOLI:
EC=4.2.2.n1 - это который выдал UniProt, но такой EC не
был найден в International Union of Biochemistry and Molecular
Biology, поэтому я использовал EC=4.2.2.1
Расшифровка кода:
| Код |
по-английски |
по-русски |
Ферментативная активность |
| 4 |
Lyases |
Лиазы |
Ферменты этого класса катализируют реакции
негидролитического отщепления и/или присоединения
функциональной группы к субстрату. При этом может
разрезаться С-С, C-N, C-O и другие связи. |
| 4.2 |
Carbon-Oxygen Lyases |
Углерод-кислородные лиазы |
Образуют и/или расщепляют
С-О связь субстрата. |
| 4.2.2 |
Acting on Polysaccharides |
Работают на полисахаридах |
Образуют и/или расщепляют связи полисахаридов. |
| 4.2.2.1 |
Hyaluronate lyase |
Гиалуронат лиаза |
Расщепляет гиалуронатные цепочки на
бета-D-GalNAc-(1->4)-бета-D-GlcA соединениях, в
итоге разрывая полисахарид на3-(4-деокси-бета-D-глюко-энуронозил)-N-ацетил-глюкозамин.
Этот фермент так же действует на хондроитин. |
Уравнение реакции:
beta-D-GalNAc-(1->4)-beta-D-GlcA = 3-(4-deoxy-beta-D-gluc-4-enuronosyl)-N-acetyl-D-glucosamine
Графическое изображение реакции:
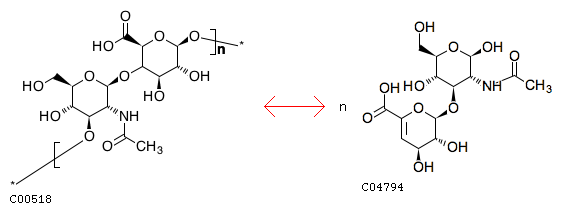
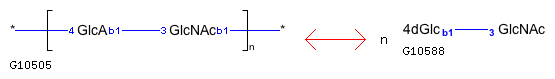
2. Метаболические пути в которых участвует фермент
MLTA_ECOLI.
Имя локуса: b2813
В БД KEGG был произведен поиск eco:b2813
По результату данного поиска был найден ген, однако указания
о его участии в метаболических путях не было.
Ссылка: http://www.genome.jp/dbget-bin/www_bget?eco:b2813
3. Поиск структурных формул в KEGG.
|
Названия
|
Идентификатор
|
Формула
|
|
L-глутамат
|
L-glutamate
|
C00025
|
|
|
L-гистидин
|
L-histidine
|
C00135
|
|
4. Посик метаболического путя от одного заданного
вещества к другому
На страничке БД
KEGG Pathway был сформирован следующий запрос:
C00025 red
C00135 green
Из выданного списка был выбран следующий путь: Histidine
metabolism. Я считаю его наиболее подходящим т.к. это
путь метаболизма гистидина, одного из данных мне веществ.
Прикрепленная карта
Как видно на данной карте существует 4 путя ферментативных
реакций перехода одного вещества в другое.
Выбранная накратчайшая цепочка ферментативных реакций:
путь: метаболизм гистидина,
цепочка: L-гистидин > L-глутамат
промежуточные соединения: Urocanate; 4-Imidazolone-5-propanote;
N-Formimino-L-glutamate и их коды соответственно C00785,
C03680, C00439
5. Сравните метаболические пути у разных организмов
|
Организм
|
Возможна ли цепочка реакций
(да/нет/неизвестно)
|
Обоснование
|
| Escherichia coli K-12 MG1655 |
нет
|
неизвестно ни одного гена необходимого
для данного процесса |
| Archaeoglobus fulgidus |
нет
|
неизвестно ни одного гена необходимого
для данного процесса |
| Arabidopsis thaliana |
нет
|
неизвестно ни одного гена необходимого
для данного процесса |
| Homo sapiens |
|
присутствуют все ферменты, необходимые
для осуществления цепочки реакций |
6. Сравнивание ферментов из далеких организмов
Мой EC код: 2.7.1.23
1. В UniProt через SRS был создан запрос (с выключенной
маской для того, чтобы поиск производился точно по такому
EC коду без вариантов с другими окончаниями после кода):
([uniprot-ECNumber:2.7.1.23*] & ([uniprot-AllText:*_HUMAN*]
| [uniprot-AllText:*_ARCFU*]))
Который нашел белки с EC-кодом у человека и археи Archaeoglobus
fulgidus (ARCFU). Всего было найдено 2 белка: NADK_HUMAN(описании:
NAD kinase) и PPNK_ARCFU (описание: Probable inorganic
polyphosphate/ATP-NAD kinase).
2. При помощи режима SW_InterProMatches для просмотра
находок, сравним доменную организацию (по PFAM) найденных
белков. У каждого из белков присутствует только по одному
и тому же домену PF01513.
Описание данного домена:
Члены данной семьи включают ATP-NAD киназы EC:2.7.1.23,
которая катализирует фосфорилирование NAD в NADP утилизируя
ATP и другие нуклеозид трифосфаты так же как и неорганический
полифосфат как источник фосфора.
Структура домена :
По страничке SRS для заданных белков переходим на ссылку
KEGG. На найденной страничке щелкнаем по кнопке "Ortholog",
а в открывшемся окне выбераем опцию Best-best (bidirectional
best hit), указав нужную группу организмов и щелкните
по кнопке "GO"
Ген ортолог NADK_HUMAN среди архей:
Организм: Methanococcus maripaludis S2: MMP1489
Ген: mmp:MMP1489
Процент идентичности: 30.9
Длина перекрывания: 304
Ген ортолог PPNK_ARCFU среди эукариот:
Организм: Paramecium tetraurelia: GSPATT00006279001
Ген: ptm:GSPATT00006279001
Процент идентичности: 29.5
Длина перекрывания: 190
В связи с тем, что организмы Археи и Эукариоты далекие,
идентичность между данными белками для определения функции
ферментов не идеальная, хотя тоже не плохая. Т.к. при
идентичности больше 40% функция ферментов сохраняется
в 90% случаев.